 Dieser Artikel ist Teil des ScienceBlogs Blog-Schreibwettbewerb 2017. Informationen zum Ablauf gibt es hier. Leserinnen und Leser können die Artikel bewerten und bei der Abstimmung einen Preis gewinnen – Details dazu gibt es hier. Eine Übersicht über alle am Bewerb teilnehmenden Artikel gibt es hier. Informationen zu den Autoren der Wettbewerbsartikel finden sich in den jeweiligen Texten.
Dieser Artikel ist Teil des ScienceBlogs Blog-Schreibwettbewerb 2017. Informationen zum Ablauf gibt es hier. Leserinnen und Leser können die Artikel bewerten und bei der Abstimmung einen Preis gewinnen – Details dazu gibt es hier. Eine Übersicht über alle am Bewerb teilnehmenden Artikel gibt es hier. Informationen zu den Autoren der Wettbewerbsartikel finden sich in den jeweiligen Texten.
——————————————————————————————————————
Digitales Erbe: DNA als Massenspeicher?
von Dennis Gregor.
Ich bin Allgemeinmediziner und interessiere mich für verschiedene wissenschaftliche Themen, auch jene, die nichts mit Medizin zu tun haben.
Um Informationen aufzubewahren, können wir sie in Stein meißeln, auf Papier schreiben, auf ein Magnetband spielen, in einem Flash-Speicher ablegen… und noch viele Dinge mehr tun, die meisten davon erst seit relativ kurzer Zeit. Viel, viel älter ist dagegen das Speichermedium, mit dem alle Lebewesen seit dem dunkelsten Archaikum ihre eigenen Baupläne aufbewahren: Die DNA speichert eine unglaubliche Menge von Informationen auf engstem Raum. Bei geeigneten Umweltbedingungen ist dieses Molekül stabil genug, um außerhalb eines Körpers tausende von Jahren zu überdauern. Könnten wir uns diese Eigenschaften zu Nutze machen und digitale Daten statt auf Festplatten auf DNA schreiben? Seit einigen Jahren gehen Wissenschaftler dieser Frage nach.
Seit Jahrtausenden bewahrt der Mensch sein Wissen, hütet es sorgsam in Büchern und füllt damit riesige Gebäude. Seit Mitte des letzten Jahrhunderts speichern wir Informationen auch auf digitalen Medien, die magnetisierbare oder optisch veränderbare Oberflächen besitzen, und sind heutzutage in der Lage, den Inhalt von tausenden von Büchern, hunderte von Stunden bewegter Bilder oder praktisch jede andere Art von Information auf einer Festplatte zu speichern, die wir bequem in unserer Jackentasche verstecken. Man könnte glauben, Speicherplatz sei kein Problem mehr.
Dabei unterschätzt man allerdings die Sammelwut unserer Spezies. Denn die Datenmengen, die die Menschheit allein im Laufe des letzten Jahrzehnts angehäuft hat, übertreffen sämtliche bisher vorhandenen Aufzeichnungen. Zwar fallen Daten in allen möglichen Bereichen an, die Hauptverursacher von Big Data findet man aber in der Forschung und in den sozialen Netzwerken. Während man am CERN die aus den Experimenten im Teilchenbeschleuniger gewonnenen Daten monatlich um ein bis zwei Petabyte anwachsen sieht, liefern große Teleskopanlagen wie das Australian Square Kilometre Array jährlich astronomische Bild- und Messdaten, die an ein Exabyte heranreichen. (Kurz zur Erinnerung für Normalverbraucher: 1 Gigabyte=109 Byte, 1 Terabyte=1012 Byte, 1 Petabyte=1015 Byte, 1 Exabyte=1018 Byte, 1 Zetabyte=1021 Byte).
Auf YouTube werden pro Minute hunderte von Stunden Video hochgeladen, die gespeichert werden wollen, und Facebook hat erst kürzlich ein neues Datenzentrum errichtet, in dem auf 6000 m2 ein Exabyte von Benutzerdaten Platz finden soll. Hochrechnungen gehen von einem weltweiten Datenuniversum von 44 Zetabyte im Jahre 2020 aus.
Das Wachstum unserer Daten übertrifft inzwischen die durch die technische Weiterentwicklung erreichte Zunahme der Speicherdichte auf magnetischen Medien. Ist es also Zeit für einen Quantensprung in der Speichertechnik, um der Datenflut Herr zu werden?
Einige Forscher haben ihren Blick in den letzten Jahren auf ein völlig andersartiges Speichermedium gerichtet. Dabei ist dieses Medium alles andere als neu, vielmehr handelt es sich um das älteste Speichersystem der Welt: Die DNA, Träger der Erbsubstanz in allen Lebewesen, wurde bereits vor etwa vier Milliarden Jahren von der Natur hervorgebracht, die sich seitdem nicht veranlasst gesehen hat, auf ein anderes Medium umzusteigen.
Die DNA (Desoxyribonukleinsäure) übertrifft bei Weitem alle unsere Speichersysteme im Hinblick auf die Packungsdichte der Information. Man vergegenwärtige sich nur einmal, dass die gesamte Erbinformation eines Menschen im Zellkern jeder einzelnen Körperzelle untergebracht ist. Zwar entspricht die dort enthaltene Information mit rund 800 Megabyte gerade einmal der Kapazität einer CD, doch wiegt die DNA, in denen sie gespeichert ist nur wenige Picogramm. Ein Exabyte, das Facebook auf einem 6000 m2 großen Gelände unterbringt, würde demnach in einem Zehntel Gramm DNA Platz finden!
Die in DNA enthaltene Information ist aber nicht nur extrem dicht gepackt, sondern bleibt unter geeigneten Bedingungen auch sehr lange erhalten. So ist es zum Beispiel gelungen, DNA aus den Knochen von Neandertalern, die vor 100.000 Jahren gelebt haben, zu extrahieren und zu entschlüsseln. Dagegen muss ein Magnetband, das gegenwärtig für die Langzeitarchivierung bevorzugte Medium, spätestens nach 30 Jahren ausgetauscht werden, um Datenverlusten vorzubeugen.
Aber wie speichert die DNA nun ihre Information? Die DNA ist ein langes fadenförmiges Molekül, das aus der Aneinanderreihung sogenannter Nucleotide hervorgeht, die in vierfacher Ausfertigung vorkommen. Ein Nucleotid besteht aus einem Zuckermolekül (der Desoxyribose), einer Phosphatgruppe und – das ist der Kniff! – einer von vier verschiedenen Basen: Adenin, Thymin, Cytosin und Guanin. In der Reihenfolge dieser Basen entlang des DNA-Fadens steckt die genetische Information, notiert in einem Alphabet mit vier Buchstaben. Obwohl der Abstand zwischen zwei Buchstaben in diesem fortlaufenden Text nur 0,34 Nanometer beträgt, ergibt das bei drei Milliarden Buchstaben in doppelter Ausführung (siehe unten) immerhin knapp zwei Meter DNA, die in jedem menschlichen Zellkern enthalten ist. Dass sie darin Platz findet, liegt daran, dass der DNA-Faden nur zwei Nanometer dick ist und in der Zelle verknäult vorliegt.
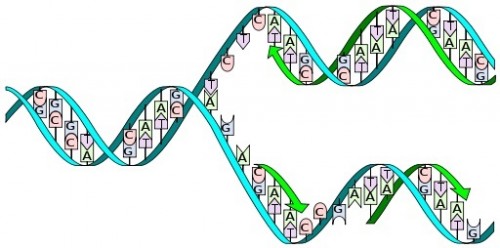
Ein weiterer Trick dieses biologischen Lexikons ist, dass es sich sehr leicht kopieren lässt. In lebenden Organismen liegt die DNA nämlich in doppelter Ausführung vor, und zwar in Form zweier parallel verlaufender Nucleotidstränge, die sich umeinander winden und die berühmte Doppelhelix bilden. Die Stränge sind dabei so zueinander orientiert, dass jeweils die Basen des einen Stranges über Wasserstoffbrücken an die Basen des anderen gekoppelt sind.
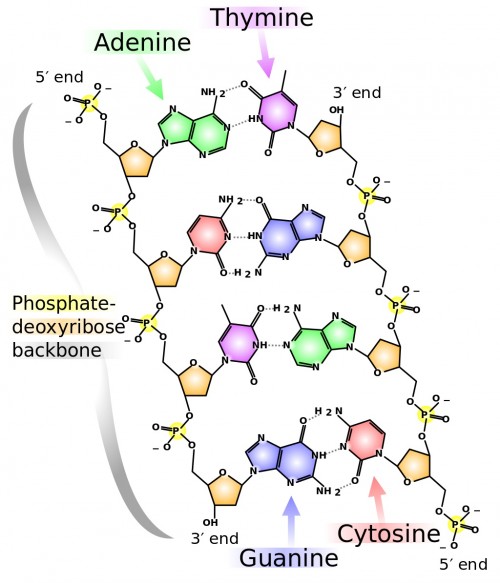
Da sich aber immer nur Adenin an Thymin und Cytosin an Guanin bindet, stellt der zweite Nucleotidstrang eine Negativkopie des ersten dar! Teilt man den DNA-Doppelstrang in seine zwei Hälften, dann kann man anhand des einen Stranges jeweils den anderen rekonstruieren. Genau das geschieht, wenn sich eine Zelle teilt und ihre DNA verdoppelt werden muss: Zuerst wird der Doppelstrang durch Enzyme gespalten und anschließend hangelt sich ein anderes Enzym, die Polymerase, an den Einzelsträngen entlang und baut jeweils die komplementären Stränge wieder auf, wobei ihr die vorhandenen Stränge das Strickmuster liefern.
Schon seit längerer Zeit sind Genforscher in der Lage, DNA nicht nur zu sequenzieren, also ihre Basenfolge zu lesen, sondern auch mittels kontrollierter chemischer Reaktionen zu synthetisieren, das heißt DNA mit einer ganz bestimmten Basenfolge künstlich herzustellen. Da liegt die Idee nicht so fern, statt biologischer Bauanleitungen einfach beliebige Daten in eine Basenfolge zu übersetzen und die entsprechende DNA zu synthetisieren. Man könnte sie dann zum Beispiel in gefriergetrocknetem Zustand lagern und zu einem späteren Zeitpunkt wieder sequenzieren um die verpackten Daten zurückzugewinnen – fertig ist die DNA-memory!
Dass dies tatsächlich funktioniert, bewiesen Forscher der Universität Harvard bereits vor fast zwanzig Jahren, indem sie eine 24 Buchstaben lange Botschaft in einem DNA-Pool mit menschlicher DNA versteckten. Offensichtlich ging es ihnen damals noch nicht um die Archivierung großer Datenmengen, sondern um die Übermittlung geheimer Nachrichten.
Knapp 15 Jahre später waren es wieder Forscher in Harvard, die sich daran wagten, nun auch die Speicherung relevanter Datenmengen auf DNA zu erproben. George Church und seine Kollegen übersetzten mehrere JPG-Bilder, ein HTML-codiertes Buch sowie ein kleines Computerprogramm, die zusammen 658 kByte auf die Waage brachten, zunächst im Computer in eine Basensequenz. Obwohl ein Nucleotid mit seinen vier möglichen Basen theoretisch zwei Bits darstellen könnte, entschieden sich die Forscher für einen „eine Base – ein Bit“ Code. Das hatte den Vorteil, dass jedes Bit durch eine von zwei verschiedenen Basen codiert werden konnte („0“ durch Adenin oder Guanin, „1“ durch Thymin oder Cytosin). Auf diese Weise ließen sich bestimmte Basenfolgen vermeiden, die bekanntermaßen bei der DNA-Synthese häufig zu Fehlern führen.
Um die DNA mit der errechneten Basensequenz herstellen und später wieder auslesen zu können, mussten die Wissenschaftler noch weitere Kunstgriffe anwenden. Zunächst einmal ist es problematisch, sehr lange DNA-Stränge zu synthetisieren, denn dabei erhöht sich die Wahrscheinlichkeit, dass falsche Nucleotide in die Kette eingefügt werden. Heute liegt das Limit schon bei deutlich über tausend Basen, vor fünf Jahren war die von Church gewählte Länge von 159 Nucleotiden realistisch. Folglich musste das Team die zu speichernden Daten auf 54.898 DNA-Schnipsel, sogenannte Oligonucleotide, verteilen, von denen jeder 96 Basen der gesamten Sequenz enthielt.
Wozu dienten aber die restlichen 63 Nucleotide in jedem Schnipsel? Einige spezielle Nucleotidsequenzen waren schlicht notwendig, um später die Kopier- und Sequenziervorgänge starten zu können. Andere dienten als Hausnummern: Während nämlich auf einer Festplatte oder auf einer CD jedes Bit seinen festen räumlichen Ort hat, an dem es stets zu finden ist, schwimmen die DNA-Schnipsel völlig ungeordnet in der Probe. Macht man sich daran, sie zu sequenzieren, um ihnen die anvertrauten Geheimnisse wieder zu entlocken, so geschieht dies in völlig zufälliger Reihenfolge. Damit man am Ende wieder intakte Dateien erhält, muss man wissen, in welcher Reihenfolge die ausgelesenen Sequenzen am Ende im Computer wieder zusammengesetzt werden sollen. Deshalb fügten die Forscher in jeden DNA-Schnipsel noch 19 Adress-Nucleotide ein, die anzeigten, an welche Position in der Gesamtsequenz das jeweilige Oligonucleotid gehörte.
Ein weiteres Problem beim Synthetisieren und Sequenzieren von DNA ist, dass beide Vorgänge fehleranfällig sind. Von 100 bis 500 Nucleotiden wird eines falsch eingebaut beziehungsweise gelesen. Nun würde man eine Festplatte, die von 500 Bytes eines verschusselt, ohne Umschweife auf dem Elektromüll deponieren. Die Harvard-Forscher stellten aber nicht nur ein einzelnes Exemplar jedes Oligonucleotids her, sondern Hunderte gleichzeitig. Sie durften erwarten, dass sich die bei der Synthese auftretenden Fehler bei jedem Exemplar an anderer Stelle befinden würden, so dass jede einzelne Basenposition bei den meisten Strängen korrekt sein würde. So waren sie in der Lage, nach dem Sequenzieren für jede Position auf den DNA-Strängen einen „Mehrheitsbeschluss“ zu errechnen. Trotz dieser massiven Mehrfachspeicherung brachten sie es auf eine Speicherdichte von 5 Petabyte pro Gramm. Allerdings verloren sie bei ihrem Experiment immer noch ganze 10 Bits – für ein Archivsystem eine inakzeptable Fehlerquote.
Inzwischen hat man auch in anderen Labors mit dem DNA-Speicher experimentiert und versucht, die Fehlerrate zu verbessern. Der nächste logische Schritt war der Einsatz eines auf mathematischen Algorithmen beruhenden Fehlerkorrektursystems. Im Prinzip wird dabei redundante, also „überflüssige“ Information abgespeichert. Dadurch können etwaige Fehler entdeckt und berichtigt werden, solange der größte Teil des Datenpaketes intakt ist. Forscher der ETH Zürich verwendeten dazu erfolgreich die sogenannte Reed-Solomon-Kodierung, die unter anderem bei der störanfälligen Funk-Kommunikation mit Raumsonden benutzt wird.
In lebenden Organismen sind es komplizierte enzymatische Reparaturmechanismen, die Mutationen in der DNA entdecken und korrigieren können. Interessanterweise verdanken wir aber unsere Existenz der Tatsache, dass diese Mechanismen nicht zu 100% effektiv sind, denn die zufälligen Erbgutveränderungen ermöglichen erst die Evolution.
Jetzt fehlt eigentlich nur noch eines, um die Datenspeicherung auf DNA wirklich praktikabel zu machen: die Möglichkeit, willkürlich auf bestimmte Datenpakete zugreifen zu können. Nach der in den früheren Experimenten von Church und anderen verwendeten Methode musste nämlich immer die gesamte in der Probe vorhandene DNA sequenziert werden, auch wenn vielleicht nur ein kleiner Teil der Information benötigt wurde. Wegen der räumlich zufälligen Verteilung der datentragenden Oligonucleotide war ein gezielter Zugriff nicht möglich.
Doch auch hier zeichnen sich Lösungen ab. Wissenschaftler der Universität Washington haben gemeinsam mit Microsoft Research im vergangenen Jahr ihr Rezept vorgestellt. Eine entscheidende Rolle spielt dabei die Polymerase-Ketten-Reaktion (PCR), mit der geringste DNA-Mengen einfach und sehr effektiv vervielfältigt werden können. Die PCR ist heute ein Routineverfahren, das in vielen Bereichen zur Anwendung kommt. Während sie in der Medizin hilft, Krankheiten durch den Nachweis von Erreger-DNA zu diagnostizieren, wird sie von Kriminalisten eingesetzt, um den genetischen Fingerabdruck eines Täters aus winzigen Blut- oder Speichelresten zu gewinnen.
Die Vervielfältigung eines DNA-Stranges läuft dabei im Prinzip ähnlich ab wie in einer lebenden Zelle: Ein Polymerase-Enzym hangelt sich am Originalstrang entlang und setzt Nucleotid für Nucleotid den gegenüberliegenden komplementären Strang zusammen, wobei es sich jeweils den passenden Baustein aus der Reaktionslösung herausfischt. Anschließend wird die Lösung erhitzt, damit sich die DNA wieder in ihre Einzelstränge aufspaltet. Dann startet von Neuem die Aktivität der Polymerase, die nun bereits zwei halbe Stränge als Vorlage vorfindet und komplementiert, so dass man zwei vollständige DNA-Doppelstränge erhält. Man sieht sofort, dass sich nun bei jedem weiteren Durchgang die Menge der DNA-Stränge verdoppelt, so dass man schnell das Wunder des exponentiellen Wachstums beobachten kann: Nach 25 Zyklen erhält man aus einem einzigen DNA-Einzelstrang 224=16 Millionen DNA-Doppelstränge.
Um diesen Vorgang anzustoßen, ist allerdings noch eine weitere Zutat erforderlich, die wir bisher unterschlagen haben, die aber gerade den Ansatzpunkt für den willkürlichen Datenzugriff bietet. Eine DNA-Polymerase kann nämlich immer nur angefangene DNA-Kopien verlängern, diese aber nicht von selbst starten. Dazu braucht sie einen primer. Das ist ein kurzes Oligonucleotid, dessen Basenfolge genau komplementär zur Anfangssequenz der DNA ist, die kopiert werden soll. Wird der primer im Überschuss zur Reaktionslösung hinzugegeben, lagert er sich von selbst an die passende Stelle am Anfang des Stranges an, der somit hier zum fertigen Doppelstrang wird. An dieser Stelle kann die Polymerase ansetzen und den Rest weiterkopieren.
Der Trick des Forscherteams bestand nun darin, außer den Adress-Nucleotiden in jeden DNA-Schnipsel noch einen weiteren kurzen Nucleotid-Code einzubauen, der als Schlüssel zu einem Paket zusammengehöriger Daten diente. Um gezielt auf ein Datenpaket zuzugreifen, synthetisierten sie viele Kopien eines zum gesuchten Schlüssel komplementären primers und starteten dann die Polymerase-Ketten-Reaktion. Auf diese Weise wurden nur die Oligonucleotide mit dem passenden Schlüssel kopiert, die am Ende in millionenfach höherer Konzentration vorlagen als die „ungewollten“ Stränge und daher selektiv sequenziert werden konnten. Mit ihrem Verfahren gelang es den Forschern, gezielt mehrere Bilddateien fehlerfrei aus dem DNA-Pool herauszulesen.
Werden wir also bald die Festplatte unseres Desktop-Computers durch PCR-Thermocycler und Sequenzierapparate ersetzen? Wann wird Facebook auf biologische Datenspeicherung umsteigen?
Abgesehen davon, dass man für den Zugriff auf die Daten etliche Stunden Zeit veranschlagen muss, dürfte der ein oder andere Speicherwillige wohl auch vom Preis des hypermodernen Archivierungssystems abgeschreckt werden. Während die Kosten für DNA-Sequenzierung aufgrund ihrer weitverbreiteten Anwendung bereits enorm gesunken sind, ist die DNA-Synthese nach wie vor sehr kostspielig. Um ein Megabyte Daten auf DNA zu schreiben, muss man rund 10.000 Euro investieren. Trotzdem sehen die Wissenschaftler, die an dem System tüfteln, Möglichkeiten für Anwendungen in naher Zukunft.
„Wenn wir (die Daten) erst einmal auf DNA geschrieben haben, kannst du sie in eine Höhle legen und vergessen, bis du sie lesen willst“, meint Nick Goldman vom European Bioinformatics Institute und hebt damit eine wesentliche Stärke des DNA-Speichers hervor. Sollen Daten, auf die nur selten zugegriffen wird, über extrem lange Zeiträume gespeichert werden, könnte sich sein Einsatz auch heute schon lohnen. Goldman rechnet vor, dass der DNA-Speicher dank fehlender Wartungskosten bei einem Speicherhorizont von 500 Jahren kosteneffizienter ist als Festplatten und Magnetbänder.
Wer seine Daten gar bis ins nächste Jahrtausend retten möchte, wittert sicher schon ein echtes Schnäppchen! Denn erst kürzlich hat man an der ETH Zürich neue Maßstäbe für die Langzeitlagerung von DNA gesetzt. Das Team um den Biochemiker Robert Grass verpackte DNA-Moleküle in winzige Glaskügelchen und ahmte damit in gewisser Weise die Konservierung von DNA in fossilen Knochen nach. Um den Gang der Jahrhunderte zu simulieren, lagerten die Forscher die DNA-Kügelchen einen Monat lang bei 70 Grad Celsius und lösten sie anschließend durch einen einfachen chemischen Prozess wieder aus ihrem Schutzmantel, um sie zu sequenzieren. Sie kamen zu dem Schluss, dass auf diese Art bei Raumtemperatur gelagerte DNA über mehr als tausend Jahre stabil wäre. Für eine Lagerung bei -18 Grad, wie sie im weltweiten Saatgut-Tresor auf Spitzbergen herrschen, errechneten sie sogar eine Haltbarkeit von einer Million Jahren!
Ob dann noch jemand da ist, der die in DNA gemeißelten Daten lesen kann oder will, ist eine andere Frage. Zumindest dürfte das know-how für die Sequenzierung von DNA dank seiner vielfältigen Anwendungen vorerst nicht aus der Mode kommen.
Bliebe noch die Frage, welche Daten wir denn mittels dieses zähen Speichersystems für die Ewigkeit aufbewahren sollen. Regierungsarchive und wichtige historische Dokumente wären mögliche Kandidaten. Grass schlägt die Memory of the World-Sammlung der UNESCO vor. Auch die Wikipedia hält er für ein interessantes Objekt, da sie einen guten Spiegel der gegenwärtigen Gesellschaft darstelle. Offenbar will er den Historikern zukünftiger Generationen die Arbeit erleichtern.

Die Optimisten unter den Bioinformatikern glauben indes, dass der biologische Speicher im Laufe eines Jahrzehnts auch für diejenigen interessant werden könnte, die vielleicht selbst noch einmal auf ihre Daten zugreifen möchten, anstatt sie lediglich für zukünftige Zivilisationen gerettet zu wissen. Denn die Kosten für DNA-Synthese und Sequenzierung sind seit ihrer Einführung um mehrere Größenordnungen gesunken und man kann wohl erwarten, dass diese Techniken aufgrund ihrer großen Bedeutung und weiten Anwendung in Medizin, Biologie und Technik in Zukunft noch billiger werden. Die Vorstellung, dass Magnetbandbibliotheken für Langzeit-backups mit der Zeit durch DNA-memory ersetzt werden, ist also keine reine Science-Fiction.
Facebook und YouTube werden aber wohl nach anderen Methoden suchen müssen, ihre Datensammelwut zu kanalisieren, schließlich leben die dort gespeicherten Inhalte nicht zuletzt von ihrer schnellen Verfügbarkeit. Irgendwann hilft vielleicht nur noch Ausmisten. Schließlich ziehen wir nicht alle fünf Jahre in eine größere Wohnung um, damit all die Dinge, die wir im Laufe der Zeit angesammelt haben, noch Platz finden, sondern machen uns in mehr oder weniger großen Abständen daran, Wichtiges von Unwichtigem zu trennen. Mancher wird sich fragen, ob all die Millionen Stunden Video, die täglich bei YouTube hochgeladen werden, unbedingt für die Nachwelt aufbewahrt werden müssen.
Literatur:
Bornholt, James, Randolph Lopez, Douglas M. Carmean, Luis Ceze, Georg Seelig, und Karin Strauss. „A DNA-Based Archival Storage System“. In Proceedings of the Twenty-First International Conference on Architectural Support for Programming Languages and Operating Systems, 637–649. ASPLOS ’16. New York, NY, USA.
Church, George M., Yuan Gao, und Sriram Kosuri. „Next-Generation Digital Information Storage in DNA“. Science 337, Nr. 6102 (28. September 2012): 1628–1628.
Extance, Andy. „How DNA could store all the world’s data“. Nature News 537, Nr. 7618 (1. September 2016): 22.
Hervorragend! Mein Favorit!
Große Klasse! Hervorragend erklärt, ein fesselndes Thema mit inspirierenden Ausblicken in die Zukunft.
Bestnote meinerseits.
Eine Idee wird zur materiellen Gewalt.
Jo, toller Artikel und auch noch was gelernt.
Wenn man so darüber nachdenkt, im Prinzip klingt das Speichern und Auslesen der DNA ja simpel, aber wie komplex es sein muss, aus einem 2 m langen Strang, der eng eingerollt ist, ein Protein auszulesen und zu replizieren, das ist für mich als Laie kaum vorstellbar, wie das funktioniert.
Und wenn das Erbgut des Menschen nur 800 MBytes ausmacht, dann wäre mit der hier beschriebenen Speichertechnik, wenn sie denn vervollkommnet würde, die Synthetisierung beliebiger echter DNA-Baupläne für Lebewesen ein Klacks (á la Jurassic Park; ja, das Zusammensuchen eines kompletten Genoms ist ein großes Problem; man könnte aber vielleicht anhand von Genomen heutiger Nachfahren und Verwandter, sprich, Vögel und Krokodile, einen Großteil der Bruchstücke wieder in die richtige Anordnung bringen; das Neandertaler-Genom hat man doch auch sequenziert, oder?).
Oder wird das weiterhin an der maximal möglichen „Schnipsellänge“ scheitern?
Spannendes Thema, sehr gut geschrieben, ein Highlight!
(Hab jetzt schon etliche Favoriten und noch nicht mal alle Artikel gelesen… )
Die Größenordnungen, um die es bei der Speicherung geht, sind übrigens echt verblüffend. Auch die lange Haltbarkeit. Ich hätte gedacht, dass die DNA unter nicht optimalen Bedingungen schneller zerfällt.
Elegant!
@Alderamin
Meines Wissens hat man DNA aus bis zu 100.000 Jahre alten Neandertalerknochen gewinnen können. Das dürfte aber wohl auch nahe der Obergrenze liegen. Viel länger können die Moleküle dann wirklich nur unter ganz speziellen künstlichen Bedingungen stabil bleiben. Bei über 60 Millionen Jahre alten Dinosaurierüberresten dürfte die Lage wohl hoffnungslos sein. (Auch wenn der Moskito, der den Saurier gestochen hat, in Bernstein eingeschlossen wurde…)
Professionell und in elegantem Stil geschrieben. Vertiefe das Thema und mach ein Buch daraus, ich würde es sofort kaufen. Danke für den interessanten Beitrag.
Ein klasse Artikel. Endlich mal nix zu meckern (meist auf hohem Niveau hier im wettbewerb :-)). Hoch interessant und klar präsentiert.
Mir fiel gleich ein sf-Roman ein, in dem die zukünftige Menschheit in ihrer „müll“-dna Infos uns Nachrichten von Vor-Millionen-Jahren enthält. 🙂 SMS der Götter quasi
@Alderamin
„das Neandertaler-Genom hat man doch auch sequenziert, oder?“
Siehe A Draft Sequence of the Neandertal Genome von et al. und Svante Pääbo (der Chef steht hier als letzter in der Autorenliste).
@Cornelia S. Gliem
Gibt es einen solchen Roman, oder hast du dir das grad spontan ausgedacht? Weil ich nämlich beim Lesen des Artikels genau dieselbe Vision für eine SF-Story hatte …
@Lercherl, Dennis
Danke, also das komplette Genom, nicht nur Teile davon (war mir nicht sicher).
Was die Saurier betrifft, ich hatte verstanden, dass die DNA auf jeden Fall kaputt geht, aber vielleicht bleiben ein paar Bruchstücke erhalten? Wenn man dann genügend viele fände und sortieren könnte… Hier wird zumindest nicht ausgeschlossen, dass man was finden könnte (ich meinte mich zu erinnern, man habe bereits Dinosaurier-DNA gefunden, aber nach Google-Recherche ging es wohl um Blutzellen ohne DNA, siehe Link).
Für in Bernstein eingeschlossene Insekten soll es auch schlecht aussehen.
Och Menno… aber wenigstens ein Mammut, bitte, bitte! 😉
@Denis Gregor
Ach das ist doch doof , wen’s an so einem Artikel so garnix zum Rummäckeln gibt. ; )
@Alderamin
Evtl. kannste irgendwann mal einen Streicheln.
https://www.nationalgeographic.de/wissenschaft/2017/08/wie-wir-das-wollhaarmammut-wieder-zum-leben-erwecken
@tomtoo
Versprechen uns Koreaner, Japaner und Russen seit wenigstens 10 Jahren, aber mehr als heiße Luft kommt da nicht. Die sollen mal hinne machen!
Das sollte sich mit ein paar Quadratmetern Kunstpelz und ein paar Litern Sekundenkleber eigentlich machen lassen.
Bleibt nur das Problem einen sehr, sehr gutmütigen Elefanten zu finden … ;-).
Auch von mir beide Daumen hoch! Sehr schöner Artikel mit klaren Ableitungen und daher leicht verständlich und in die Tiefe gehend zugleich. Bisher mein Favorit!
@PDP10
Wenn das gutmütige Tier nach der Prozedur mit dem Sekundenkleber und dem Kunstpelz anschließend auch noch stilgerecht von Afrika nach Sibirien versetzt wird, wird es sicher endgültig die Nase voll haben… 😉
Aber wir schweifen ab.
@tomtoo
Von der Arbeit von Church hatte ich noch nicht gehört. Hier ein kritischer Artikel der BBC, der auch das Für und Wider betrachtet. Ja, irgendwann wird es gelingen, aber es wird noch einige Zeit dauern, und die Motivation ist eigentlich eine ganz andere, als man denkt.
Bilder als DNS-Sequenzen speichern … wurde schon mal versucht/gemacht aus einem DNS-Strang (Fruchtfliege, Mensch …) ein Bild zu generieren?
Klasse Artikel. Kurz, Prägnant und Interessant. Bisher mein absoluter Favorit!
@Alderamin:
Das wäre doch mal ein toller Artikel für den nächsten Wettbewerb. *notizkritzel*
Auch spannend fände ich, wie man die Wikipedia so in DNA codieren kann, dass sie für zukünftige Zivilisationen lesbar bleibt. Zeichencodierung (ASCII, UTF-8 CP1250 etc.) ist ja schon zu unseren Lebzeiten ein Albtraum für jeden Datenbankadmin. Da braucht es eine gute und haltbare Anleitung 😉
Wahrscheinlich, Dampier, zuerst fiel mir allerdings dieser Erzählstrang in Star Trek ein, bei dem die Urahnen der vielen über den Quadranten verstreuten Gattungen irgendwelche Hinweise auf sich selber genau so kodierten.
Witzigerweise hat mir vor Jahren einmal ein Studienkollege erzählt, er hätte eine Idee für eine science fiction story, in der irgendwelche Zukunftswesen andere Wesen als Haustiere züchteten, nur um in deren DNA ihre Daten zu speichern. Das war übrigens zu Zeiten, als 800MB noch eine Menge Holz waren. Wenn in dieser Welt dann die Herrenwesen ausstürben, hätte man das genannte Szenario: die anderen Wesen könnten irgendwann geheimnisvolle Daten in ihrer Müll-DNA entdecken.
Soweit die Vorlage. Wer macht den Roman draus…?
Man kann natürlich die DNA Sequenz von jedem (sequenzierten) Organismus als Bild darstellen. Das gibt aber einfach nur Rauschen. In bestimmten, repetitiven Bereichen des Erbguts bekommt man vielleicht auch lustige Muster, aber das wars dann auch.
Was aber kürzlich gemacht wurde ist, in der DNA eines Bakteriums einen Film zu speichern und wieder auszulesen.
Komplexes Thema verständlich gemacht und dann auch noch so geschrieben, daß man die Lust am Weiterlesen an keiner Stelle verliert. Eine Eins mit Sternchen.
Wow.
Ein Thema (zuverlässig langfristige Datenspeicherung in zumindest aktuell ausreichender Menge), das mich schon mein gesamtes Berufsleben (IT-Spezi) leidenschaftlich verfolgte (und bislang zu meinem Mißfallen nie befriedigend gelöst wurde), so dermaßen spannend und knackig darzustellen:
Ich ziehe meinen (nicht vorhandenen) Hut. Trotz enger Konkurrenz bisher mein absoluter Favorit! Überzeugend und gut nachvollziehbar dargelegt!
Ach ja, und nochmal ein Sternchen dafür, daß Allgemeinmediziner im Allgemeinen(!) wesentlich mehr können als Hömeo-i-was zu vertreiben. Sondern richtig was drauf haben (können), kenne ich ja zumindest schon mal vereinzelt (dafür auch Dank an nnF und einige andere Ärzte, Pharmazeuten und Apotheker hier). Danke dafür.
Ihr macht mir wieder Hoffnung …
Und INH & Co natürlich auch!
Ein ausgezeichnetes Blog! Vermutlich hat der „Allgemeinmediziner“ „Das autistisch-undisziplinierte Denken in der Medizin und seine Überwindung“ von Bleuler gelesen.
Dennis kam mir schon in Kommentar #22 zuvor.
Eine perverse Idee wäre es, wenn man selbst generierte Daten-DNS-Stränge ins Erbgut seiner eigenen Kinder oder in Pflanzen oder Tiere einschleust. Da sich die Datenstränge mit jeder Generation weitervererben, erhielte man sehr viele Sicherheitskopien.
Florian Freistetters Artikel hat mir gut gefallen. Als Langzeitspeicher wichtiger Daten auf kleinstem Raum wäre das Verfahren der DNS-Synthese sicherlich geeignet. Für den täglichen Gebrauch allerdings zu umständlich.
@StefanL
Ich würde wetten, dass sich Leute schon solche „Bilder“ angeschaut haben. Es gibt auch ne Menge Leute, die sich Daten auf Audiokassetten angehört haben und das irgendwie gemütlich fanden. Beides hat ungefähr gleich viel Sinn. :o)
@Dennis: Kurz stockte ich beim Wort „Quantensprung“, war sogar etwas genervt von dessen Anwendung. Zum Glück sah ich darüber hinweg und las weiter. Geiles Thema, spannend geschrieben. Danke.
@René: Entschuldige meine Verwendung dieses abgelutschten Ausdrucks. Freue mich, dass Du trotzdem weitergelesen hast!
@Bernhard Kletzenbauer: Der Artikel ist nicht von Florian Freistetter geschrieben, sondern ist ein Beitrag zu einem von ihm organisierten Schreibwettbewerb. Aber diese Verwirrung gab es hier schon gelegentlich… 🙂
@Dennis, #32
Meiner Meinung nach ist das nicht verwunderlich, wenn man sich mal die Seitenaufteilung ansieht.
Ganz oben steht:
StartseiteAstrodicticum Simplex
Digitales Erbe: DNA als Massenspeicher?
Von Florian Freistetter / 10. Oktober 2017 / 32 Kommentare
Erst ein Stück weiter unten steht:
Digitales Erbe: DNA als Massenspeicher?
von Dennis Gregor.
Ich bin Allgemeinmediziner und…
Wenn ich als Kommentarschreiber mal auf die Schnelle nachlese, wer den Artikel verfaßt hat, finde ich ganz oben den Namen Florian Freistetter und habe keine Veranlassung noch nach einem anderen Namen zu suchen.
@Bernhard: „Wenn ich als Kommentarschreiber mal auf die Schnelle nachlese,“
Ich bin halt Optimist und gehe davon aus das Leute die Kommentare schreiben auch tatsächlich die Artikel vorher lesen. Und wenn man das tut, dann kann man schwerlich übersehen das es sich um einen Gastbeitrag handelt der nicht von mir stammt…
@Florian Freistetter, #34
*rolleyes*
Wenn ich nachlese, dann habe ich den Artikel logischerweise vorher auch gelesen.
Ich war der Meinung, daß ich in Kommentar #33 eindeutig beschrieben habe, wie es zu den Namensfehlern kommen kann.
Ich finde den Artikel sehr gut, wollte dir nur noch einige Tipps auf den Weg geben. Die ersten drei Absätze können eigentlich weg oder zusammengefasst werden. Diese Stimmungssetting ist nicht so meins und packt den Leser üblicherweise nicht.
Du hast sehr gut erklärt, aber meiner Erachstens könntest du versuchen, auf allzuviele Fachbegriffe zu verzichten. Schon bei DNA fängt es bei vielen an – die science blogs Leser sind das gewöhnt, aber sobald du aus der Bubble draußen bist…
Dennoch, gut gemacht!
[…] Platz 1: Digitales Erbe: DNA als Massenspeicher? […]